2020年4月2日,山東農業大學作物生物學國家重點實驗室/農學院教授、博士生導師曾范昌課題組,在國際知名植物科學期刊《The Plant Journal》(SCI生物學一區)在線發表了題為“Ethyl methanesulfonate (EMS) mutant library construction in G. hirsutum L. for allotetraploid functional genomics and germplasm innovation”的最新研究進展。

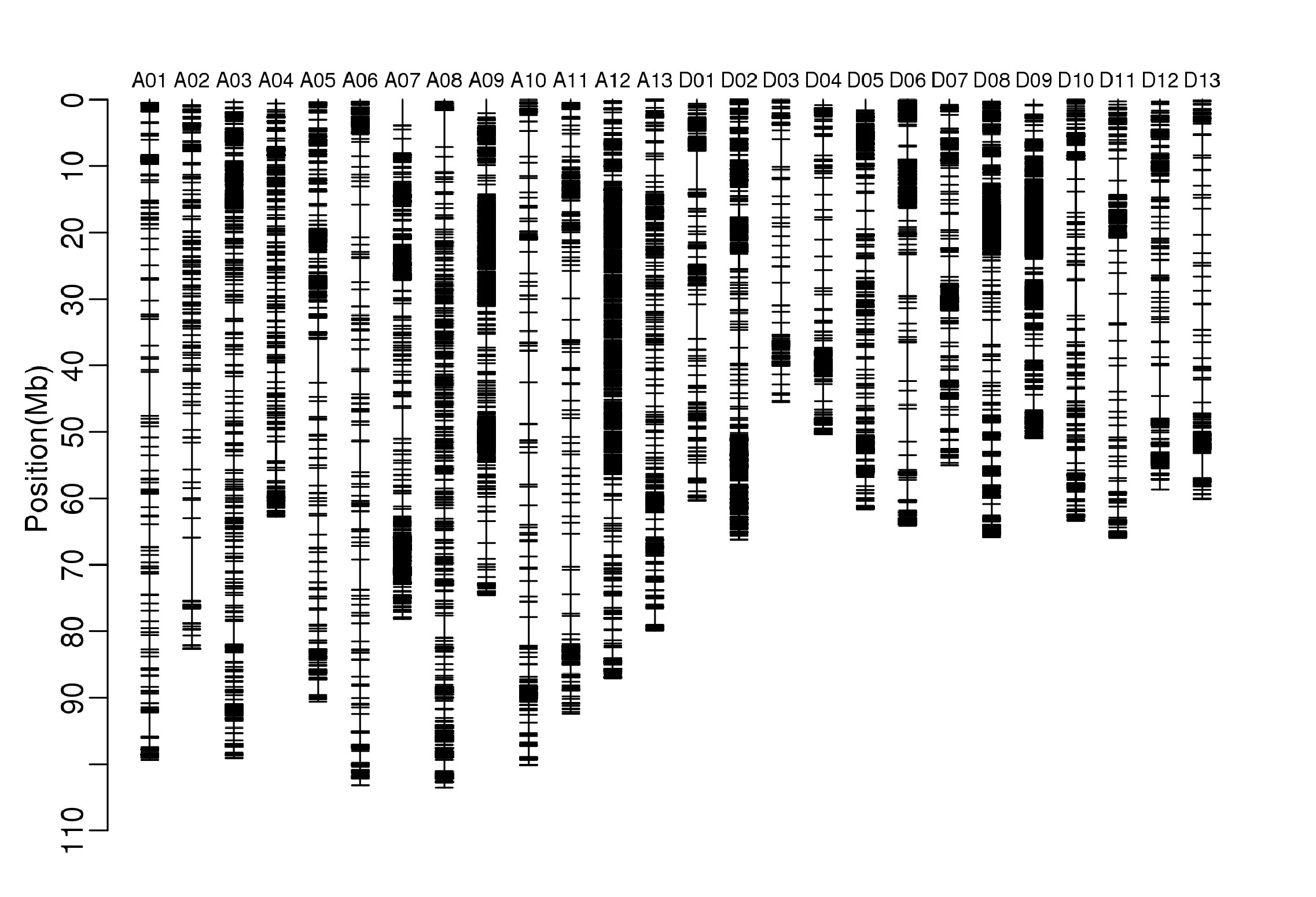
基諾賽克利用重測序方法,助力曾范昌團隊進行棉花EMS處理的突變體文庫中的全基因組突變密度檢查和質量評估。結果顯示,EMS誘導的SNP通常以理想的突變均勻性分布在棉花突變體基因組中。棉花突變體的26條染色體上的全基因組EMS SNP計數和密度顯示,共有65,286個EMS SNP突變,突變密度高(每Mb 37.76)。該研究提供了用于基于EMS的突變文庫構建的基本實用參考指南,特別是在異源四倍體作物中。更重要的是,突變文庫是進行復雜的異源四倍體性狀基礎研究以及棉花育種的寶貴資源。
基諾賽克定位于國內領先的群體遺傳分析與基因分型檢測供應商,在群體遺傳分析方面,通過簡化基因組測序、基因組重測序、混池關聯分析法(BSA)測序、Target-seq、真核轉錄組測序等手段,為廣大客戶提供超高性價比的解決方案;在基因分型檢測方面,基諾賽克專注于全基因組SNP標記Panel開發、高通量SNP基因分型、SSR標記(毛細管/常規電泳)、農作物品種指紋鑒定等,提供讓廣大客戶“看得見、摸得著、用得起”的技術和服務。公司技術團隊經驗豐富,曾參與水稻、玉米、花生、油菜、棉花、大豆、小麥、大麥、馬鈴薯、芝麻、芥菜、蘿卜、大蒜、茶樹、藍莓、枸杞、黃蓮木等植物以及牛、豬等大型動物基因組學項目,可為客戶提供專業、合理、高性價比的科研服務。在生物信息分析方面,基諾賽克擁有豐富的項目經驗,可根據不同的課題需求,提供個性化的深度數據挖掘服務,為廣大客戶在群體遺傳研究以及全基因組分子標記選擇與應用方面提供解決方案。



